Ingenieros recrean 500 millones de años de evolución con IA y logran una proteína fluorescente innovadora
2025-01-21
Autor: José
Innovaciones en el diseño de proteínas
Recientemente, el Premio Nobel de Química fue otorgado a David Baker por su trabajo en el diseño computacional de proteínas, y a Demis Hassabis y John M. Jumper por sus contribuciones en la predicción de estructuras proteicas. Estas innovaciones han abierto nuevas puertas en el entendimiento y manipulación de proteínas, un camino que continúan explorando varios laboratorios a nivel global.
Creación de una proteína fluorescente artificial
Entre estos laboratorios, la empresa estadounidense EvolutionaryScale ha logrado avanzar significativamente al crear una proteína fluorescente artificial. Este avance se basa en la simulación de 500 millones de años de evolución natural, una hazaña que combina ciencia y tecnología de punta.
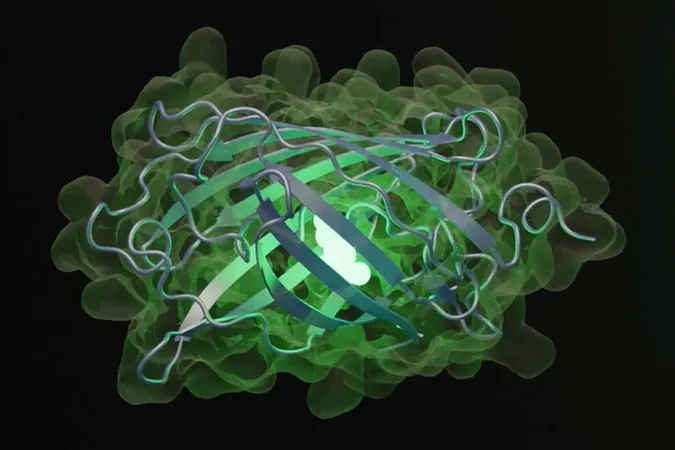
Características de la proteína esmGFP
La nueva proteína, que han nombrado esmGFP, es parte de la familia de proteínas fluorescentes verdes (GFP), que se encuentran en la naturaleza, como en algunas medusas. Cabe destacar que el descubrimiento original de estas proteínas floreció en 2008 con la obtención del Premio Nobel.
A diferencia de las proteínas GFP naturales, la esmGFP presenta una estructura que combina elementos de las GFP conocidas mientras incorpora características únicas. Aunque no hay evidencias de que esta proteína artificial exista en la naturaleza, su proyección nos permite vislumbrar alternativas evolutivas que podrían haber surgido en un universo paralelo.
Uso de modelos generativos en la creación de proteínas
El equipo detrás de este avance utilizó un modelo generativo llamado ESM3 (EvolutionaryScale Model 3), diseñado específicamente para la creación de proteínas en lugar de simplemente generar texto, como su nombre podría sugerir. Este innovador modelo analiza combinaciones infinitas de secuencias, estructuras tridimensionales y funcionalidades de proteínas. Esta capacidad es crucial, ya que tanto la secuencia de aminoácidos como la forma en que se pliegan afectan directamente la función de las proteínas, algo que los modelos anteriores no podían evaluar de manera efectiva.
Entrenamiento del modelo generativo
Para llevar a cabo este ambicioso proyecto, el equipo necesitó entrenar su modelo con más de 771.000 millones de paquetes de datos, provenientes de 3.150 millones de secuencias de proteínas, 236 millones de estructuras, y 539 millones de proteínas con sus correspondientes funciones. Los resultados obtenidos se han publicado en la revista Science, abriendo un nuevo capítulo en la investigación proteica.
Implicaciones médicas y futuras aplicaciones
La creación de estas proteínas que nunca existieron puede no solo alimentarnos con fascinantes reflexiones sobre lo que pudo haber sido, sino que también tiene aplicaciones prácticas en el ámbito de la medicina. Identificar nuevas proteínas que imiten las funciones de las proteínas naturales puede ser clave en la lucha contra numerosos trastornos de salud.
El futuro de la biotecnología
Esto representa un hito en el diseño de proteínas y ofrece un vistazo emocionante hacia el futuro de la biotecnología. Al respecto, también se han evidenciado otros desarrollos, como en el caso de una planta de tomate que produce proteínas fluorescentes para indicar a los agricultores necesidades críticas de riego o situaciones de peligro.
 Brasil (PT)
Brasil (PT)
 Canada (EN)
Canada (EN)
 Chile (ES)
Chile (ES)
 Česko (CS)
Česko (CS)
 대한민국 (KO)
대한민국 (KO)
 España (ES)
España (ES)
 France (FR)
France (FR)
 Hong Kong (EN)
Hong Kong (EN)
 Italia (IT)
Italia (IT)
 日本 (JA)
日本 (JA)
 Magyarország (HU)
Magyarország (HU)
 Norge (NO)
Norge (NO)
 Polska (PL)
Polska (PL)
 Schweiz (DE)
Schweiz (DE)
 Singapore (EN)
Singapore (EN)
 Sverige (SV)
Sverige (SV)
 Suomi (FI)
Suomi (FI)
 Türkiye (TR)
Türkiye (TR)